4 Entropie
L’entropie est la surprise moyenne apportée par l’observation des individus d’une communauté, d’autant plus grande qu’un individu appartient à une espèce plus rare. L’entropie HCDT permet d’unifier les indices classiques de diversité : son paramètre, appelé ordre, fixe l’importance donnée aux espèces rares. L’entropie d’ordre 0 est la richesse ; celle d’ordre 1, l’indice de Shannon ; celle d’ordre 2, celui de Simpson. L’entropie est la moyenne du logarithme déformé de la rareté des espèces, définie comme l’inverse de leur probabilité.
L’entropie va de pair avec la diversité au sens strict (Nombres de Hill) : le nombre d’espèces équiprobables dont l’entropie est la même que celle de la communauté réelle. La diversité est l’exponentielle déformée de l’entropie. Les profils de diversité représentent la diversité en fonction de son ordre et permettent la comparaison de communautés.
L’estimation de la diversité est difficile pour des ordres inférieurs à \(0,5\) dans des taxocènes très divers comme les arbres des forêts tropicales.
L’entropie peut être entendue comme la surprise moyenne fournie par l’observation d’un échantillon. C’est intuitivement une bonne mesure de diversité (Pielou 1975). Ses propriétés mathématiques permettent d’unifier les mesures de diversité dans un cadre général.
4.1 Définition de l’entropie
Les textes fondateurs sont Davis (1941) et surtout Theil (1967) en économétrie, et Shannon (1948; 1963) pour la mesure de la diversité. Une revue est fournie par Maasoumi (1993).
Considérons une expérience dont les résultats possibles sont \(\{r_1, r_2, \dots , \ r_S\}\). La probabilité d’obtenir \(r_s\) est \(p_s\), et \(\mathbf{p}=(p_1, p_2, \dots, p_S)\) est le vecteur composé des probabilités d’obtenir chaque résultat. Les probabilités sont connues a priori. Tout ce qui suit est vrai aussi pour des valeurs de \(r\) continues, dont on connaîtrait la densité de probabilité.
On considère maintenant un échantillon de valeurs de \(r\). La présence de \(r_s\) dans l’échantillon est peu étonnante si \(p_s\) est grande : elle apporte peu d’information supplémentaire par rapport à la simple connaissance des probabilités. En revanche, si \(p_s\) est petite, la présence de \(r_s\) est surprenante. On définit donc une fonction d’information, \(I(p_s)\), décroissante quand la probabilité augmente, de \(I(0) > 0\) (éventuellement \(+\infty\)) à \(I(1) = 0\), parce qu’observer un résultat certain n’apporte aucune information. Chaque valeur observée dans l’échantillon apporte une certaine quantité d’information, dont la somme est l’information de l’échantillon. Patil and Taillie (1982) appellent l’information “rareté”.
La quantité d’information attendue de l’expérience est \(\sum^S_{s=1}{p_s I(p_s) = H(\mathbf{p})}\). Si on choisit \(I(p_s) = -\ln(p_s)\), \(H(\mathbf{p})\) est l’indice de Shannon, mais bien d’autres formes de \(I(p_s)\) sont possibles. \(H(\mathbf{p})\) est appelée entropie. C’est une mesure de l’incertitude (de la volatilité) du résultat de l’expérience. Si le résultat est certain (une seule valeur \(p_S\) vaut 1), l’entropie est nulle. L’entropie est maximale quand les résultats sont équiprobables.
Si \(\mathbf{p}\) est la distribution des probabilité des espèces dans une communauté, Patil and Taillie (1982) montrent que :
- Si \(I(p_s) = (1 - p_s) / {p_s}\), alors \(H(\mathbf{p})\) est le nombre d’espèces \(S\) moins 1 ;
- Si \(I(p_s) = -\ln(p_s)\), alors \(H(\mathbf{p})\) est l’indice de Shannon ;
- Si \(I(p_s) = 1 - p_s\), alors \(H(\mathbf{p})\) est l’indice de Simpson.
Ces trois fonctions d’information sont représentées en figure ??.
Le code R nécessaire pour réaliser la figure est :
I0 <- function(p) (1- p) / p
I1 <- function(p) -log(p)
I2 <- function(p) 1 - p
tibble(x = c(0, 1)) %>%
ggplot(aes(x)) +
stat_function(fun = I0) +
stat_function(fun = I1, lty = 2) +
stat_function(fun = I2, lty = 3) +
coord_cartesian(ylim = c(0, 10)) +
labs(x = "p", y = "I(p)")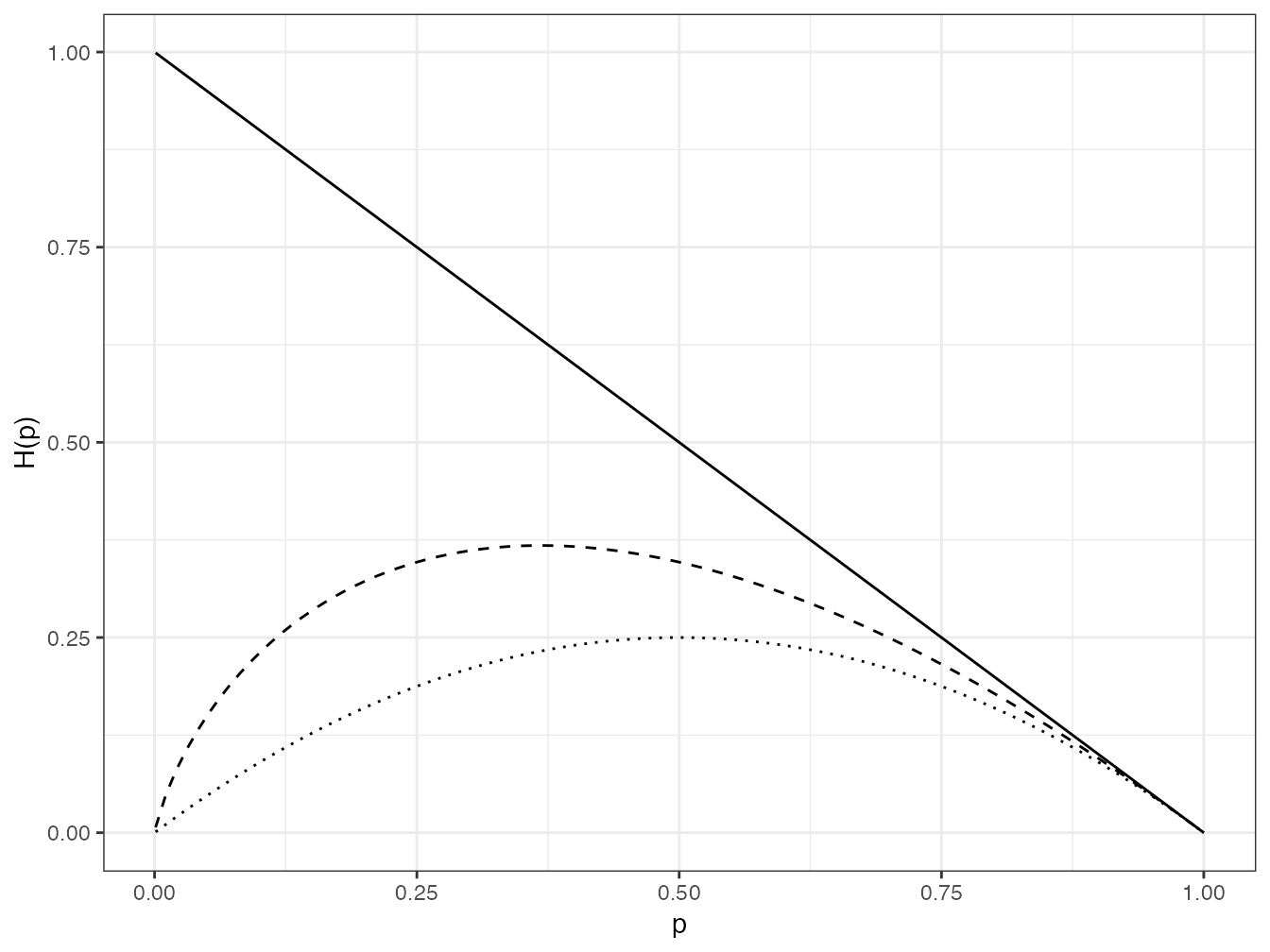
Figure 4.1: Valeur de \(p_{s}I(p_s)\) dans le nombre d’espèces (trait plein), l’indice de Shannon (pointillés longs) et l’indice de Simpson (pointillés). Les espèces rares contribuent peu, sauf pour le nombre d’espèces.
La contribution de chaque espèce à la valeur totale de l’entropie est représentée figure 4.1.
Code R :
4.2 Entropie relative
Considérons maintenant les probabilités \(q_s\) formant l’ensemble \(\mathbf{q}\) obtenues par la réalisation de l’expérience. Elles sont différentes des probabilités \(p_s\), par exemple parce que l’expérience ne s’est pas déroulée exactement comme prévu. On définit le gain d’information \(I(q_s, p_s)\) comme la quantité d’information supplémentaire fournie par l’observation d’un résultat de l’expérience, connaissant les probabilités a priori. La quantité totale d’information fournie par l’expérience, \(\sum^S_{s=1}{q_s I(q_s, p_s)} = H(\mathbf{q}, \mathbf{p})\), est souvent appelée entropie relative. Elle peut être vue comme une distance entre la distribution a priori et la distribution a posteriori. Il est possible que les distributions \(\mathbf{p}\) et \(\mathbf{q}\) soit identiques, que le gain d’information soit donc nul, mais les estimateurs empiriques n’étant pas exactement égaux entre eux, des tests de significativité de la valeur de \(\hat{H}(\mathbf{q},\mathbf{p})\) seront nécessaires.
Quelques formes possibles de \(H(\mathbf{q}, \mathbf{p})\) sont :
- La divergence de Kullback-Leibler (Kullback and Leibler 1951) connue par les économistes comme l’indice de dissimilarité de Theil (1967) : \[\begin{equation} \tag{4.1} T = \sum^S_{s=1}{q_{s}\ln\frac{q_s}{p_s}}; \end{equation}\]
- Sa proche parente, appelée parfois deuxième mesure de Theil (Conceição and Ferreira 2000), qui inverse simplement les rôles de \(p\) et \(q\) : \[\begin{equation} \tag{4.2} L = \sum^S_{s=1}{p_{s}\ln\frac{p_s}{q_s}}. \end{equation}\]
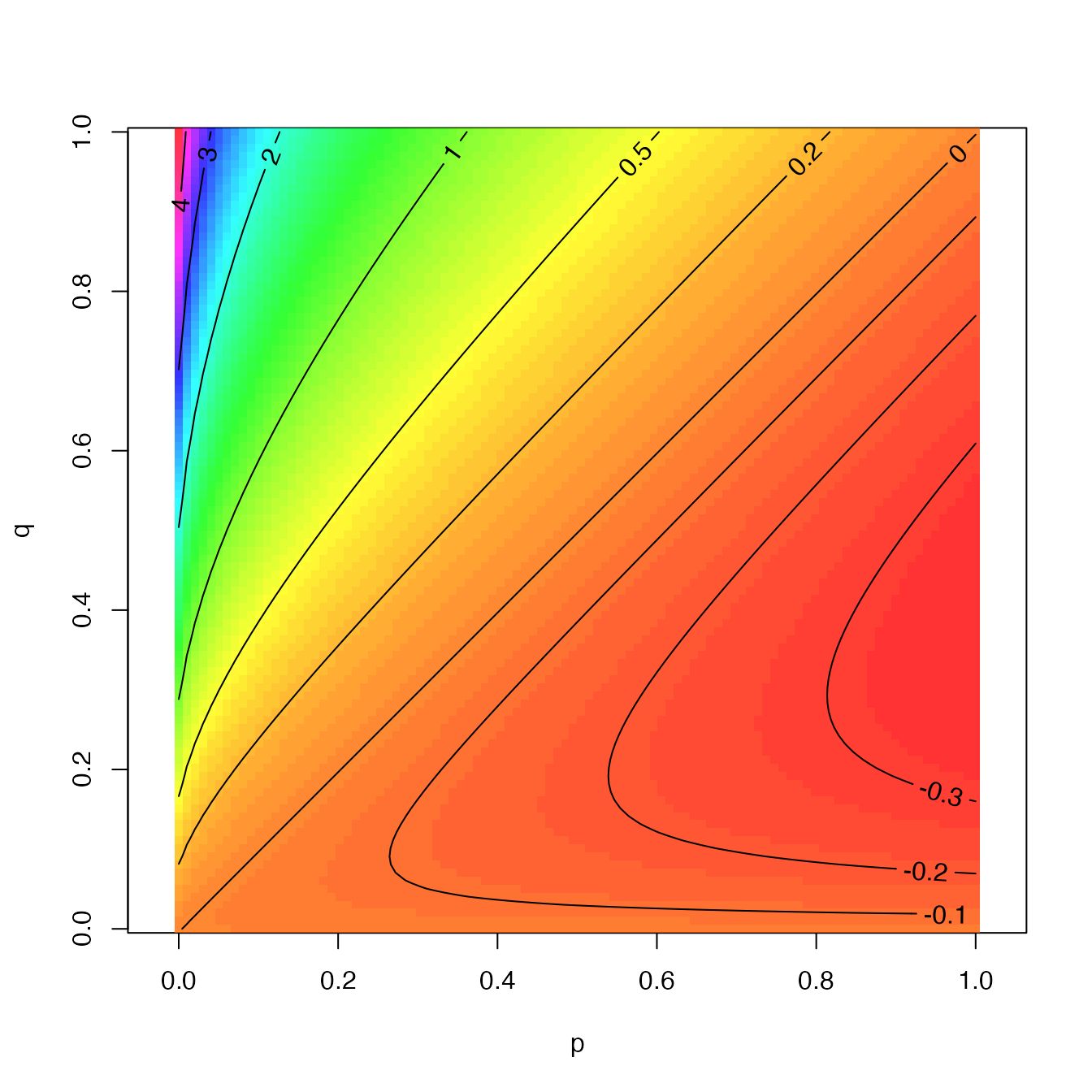
Figure 4.2: Valeur de \(q \ln(q / p)\) en fonction de \(p\) et \(q\). La divergence de Kullback-Leibler est la somme de cette valeur pour toutes les espèces
L’entropie relative est essentielle pour la définition de la diversité \(\beta\) présentée dans le chapitre ??. En se limitant à la diversité \(\alpha\), on peut remarquer que l’indice de Shannon est la divergence de Kullback-Leibler entre la distribution observée et l’équiprobabilité des espèces (Marcon et al. 2012).
La contribution à la divergence de chaque couple \((p_s, q_s)\) est représentées en figure 4.2. Elle est positive quand \(q_s > p_s\) (la valeur observée est plus grande que la valeur attendue), et croît avec \(q_s\) pour \(p_s\) fixé. Elle tend vers l’infini quand \(p_s \to 0\). Les valeurs les plus négatives ne sont pas obtenues pour les valeurs minimales de \(q\) parce que les évènements très rarement observés influent peu sur la quantité d’information totale. Le minimum \(-e^{-1} \approx 0.37\) est atteint pour \(p_s = 1\) et \(q_s = e^{-1}\), la valeur qui annule la dérivée de \(q \ln(q)\). En somme, la divergence de Kullback-Leibler est surtout influencée par les évènements beaucoup plus observés qu’attendus.
Le code R nécessaire pour réaliser la figure est :
4.3 L’appropriation de l’entropie par la biodiversité
MacArthur (1955) est le premier à avoir introduit la théorie de l’information en écologie (Ulanowicz 2001). MacArthur s’intéressait aux réseaux trophiques et cherchait à mesurer leur stabilité : l’indice de Shannon qui comptabilise le nombre de relations possibles lui paraissait une bonne façon de l’évaluer. Mais l’efficacité implique la spécialisation, ignorée dans \(H\) qui est une mesure neutre (toutes les espèces y jouent le même rôle). MacArthur a abandonné cette voie.
Les premiers travaux consistant à généraliser l’indice de Shannon sont dus à Rényi (1961). L’entropie d’ordre \(q\) de Rényi est
\[\begin{equation} ^{q}\!R =\frac{1}{1 - q \ln\sum^S_{q=1}{p^q_s}}. \end{equation}\]
Rényi pose également les axiomes pour une mesure d’entropie \(R(\mathbf{p})\), où \(\mathbf{p}=(p_1, p_2, \dots, p_S)\) :
- La symétrie : les espèces doivent être interchangeables, aucune n’a de rôle particulier et leur ordre est indifférent ;
- La mesure doit être continue par rapport aux probabilités ;
- La valeur maximale est atteinte si toutes les probabilités sont égales.
Il montre que \(^{q}\!R\) respecte les 3 axiomes.
Patil and Taillie (1982) ont montré de plus que :
- L’introduction d’une espèce dans une communauté augmente sa diversité (conséquence de la décroissance de \(g(p_s)\)) ;
- Le remplacement d’un individu d’une espèce fréquente par un individu d’une espèce plus rare augmente l’entropie à condition que \(R(\mathbf{p})\) soit concave. Dans la littérature économique sur les inégalités, cette propriété est connue sous le nom de Pigou-Dalton (Dalton 1920).
Hill (1973) transforme l’entropie de Rényi en nombres de Hill, qui en sont simplement l’exponentielle :
\[\begin{equation} \tag{4.3} ^{q}\!D = {\left(\sum^S_{s=1}{p^q_s}\right)}^{\frac{1}{1 - q}}. \end{equation}\]
Le souci de Hill était de rendre les indices de diversité intelligibles après l’article remarqué de Hurlbert (1971) intitulé “le non-concept de diversité spécifique”. Hurlbert reprochait à la littérature sur la diversité sa trop grande abstraction et son éloignement des réalités biologiques, notamment en fournissant des exemples dans lesquels l’ordre des communautés n’est pas le même selon l’indice de diversité choisi. Les nombres de Hill sont le nombre d’espèces équiprobables donnant la même valeur de diversité que la distribution observée. Ils sont des transformations simples des indices classiques :
- \(^{0}\!D\) est le nombre d’espèces ;
- \(^{1}\!D = e^H\), l’exponentielle de l’indice de Shannon ;
- \(^{2}\!D = {1} / {(1- E)}\), l’inverse de l’indice de concentration de Simpson, connu sous le nom d’indice de Stoddart (1983).
Ces résultats avaient déjà été obtenus avec une autre approche par MacArthur (1965) et repris par Adelman (1969) dans la littérature économique.
Les nombres de Hill sont des “nombres effectifs” ou “nombres équivalents”. Le concept a été défini rigoureusement par Gregorius (1991), d’après Wright (1931) (qui avait le premier défini la taille effective d’une population) : étant donné une variable caractéristique (ici, l’entropie) fonction seulement d’une variable numérique (ici, le nombre d’espèces), dans un cas idéal (ici, l’équiprobabilité des espèces), le nombre effectif est la valeur de la variable numérique pour laquelle la variable caractéristique est celle du jeu de données.
Gregorius (2014) montre que de nombreux autres indices de diversité sont acceptables dans le sens où ils vérifient les axiomes précédents et, de plus, que la diversité d’un assemblage de communautés est obligatoirement supérieure à la diversité moyenne de ces communautés (l’égalité n’étant possible que si les communautés sont toutes identiques). Cette dernière propriété sera traitée en détail dans la partie consacrée à la décomposition de la diversité. Ces indices doivent vérifier deux propriétés : leur fonction d’information doit être décroissante, et ils doivent être une fonction strictement concave de \(p_s\). Parmi les possibilités, \(I(p_s) = \cos{(p_s \pi / 2)}\) est envisageable par exemple : le choix de la fonction d’information est virtuellement illimité, mais seules quelques unes seront interprétables clairement.
Un nombre équivalent d’espèces existe pour tous ces indices, il est toujours égal à l’inverse de l’image de l’indice par la réciproque de la fonction d’information :
\[\begin{equation} \tag{4.4} D = \frac{1}{I^{-1} \left( \sum^S_{s=1}{p_s I(p_s)} \right)}. \end{equation}\]
D’autres entropies ont été utilisées, avec plus ou moins de succès. Par exemple, Ricotta and Avena (2003) proposent d’utiliser la fonction d’information \(I(p_s) = -\ln(k_s)\) où \(k_s\) est la dissimilarité totale de l’espèce \(s\) avec les autres (par exemple, la somme des distances aux autres espèces dans un arbre phylogénétique, voir section ??), normalisée pour que \(\sum_s{k_s} = 1\). Ainsi, les espèces les plus originales apportent peu d’information, ce qui n’est pas très intuitif. Les auteurs montrent que leur mesure est la somme de l’entropie de Shannon et de la divergence de Kullback-Leibler entre les probabilités et les dissimilarités des espèces. Ricotta and Szeidl (2006) ont défini plus tard une entropie augmentant avec l’originalité de chaque espèce, présentée au chapitre ??.
4.4 Entropie HCDT
Tsallis (1988) propose une classe de mesures appelée entropie généralisée, définie par Havrda and Charvát (1967) pour la première fois et redécouverte plusieurs fois, notamment par Daróczy (1970), d’où son nom entropie HCDT (voir Mendes et al. (2008), page 451, pour un historique complet) :
\[\begin{equation} \tag{4.5} ^{q}\!H = \frac{1}{q-1} \left( 1 - \sum^S_{s=1}{p^q_s} \right). \end{equation}\]
Tsallis a montré que les indices de Simpson et de Shannon étaient des cas particuliers d’entropie généralisée, retrouvant, sans faire le rapprochement (Ricotta 2005a), la définition d’un indice de diversité de Patil and Taillie (1982).
Ces résultats ont été complétés par d’autres et repris en écologie par Keylock (2005) et Jost (2006, 2007). Là encore :
- Le nombre d’espèces moins 1 est \(^{0}\!H\) ;
- L’indice de Shannon est \(^{1}\!H\) ;
- L’indice de Gini-Simpson est \(^{2}\!H\).
L’entropie HCDT est particulièrement attractive parce que sa relation avec la diversité au sens strict est simple, après introduction du formalisme adapté (les logarithmes déformés). Son biais d’estimation peut être corrigé globalement, et non seulement pour les cas particuliers (nombre d’espèces, Shannon, Simpson). Enfin, sa décomposition sera présentée en détail dans le chapitre ??.
4.5 Logarithmes déformés
L’écriture de l’entropie HCDT est largement simplifiée en introduisant le formalisme des logarithmes déformés (Tsallis 1994). Le logarithme d’ordre \(q\) est défini par \[\begin{equation} \tag{4.6} \ln_q{x} = \frac{x^{1-q} - 1}{1 - q}, \end{equation}\] dont la forme est identique à la transformation de Box and Cox (1964) utilisée en statistiques pour normaliser une variable.
Le logarithme déformé converge vers le logarithme naturel quand \(q \to 1\) (figure 4.3).
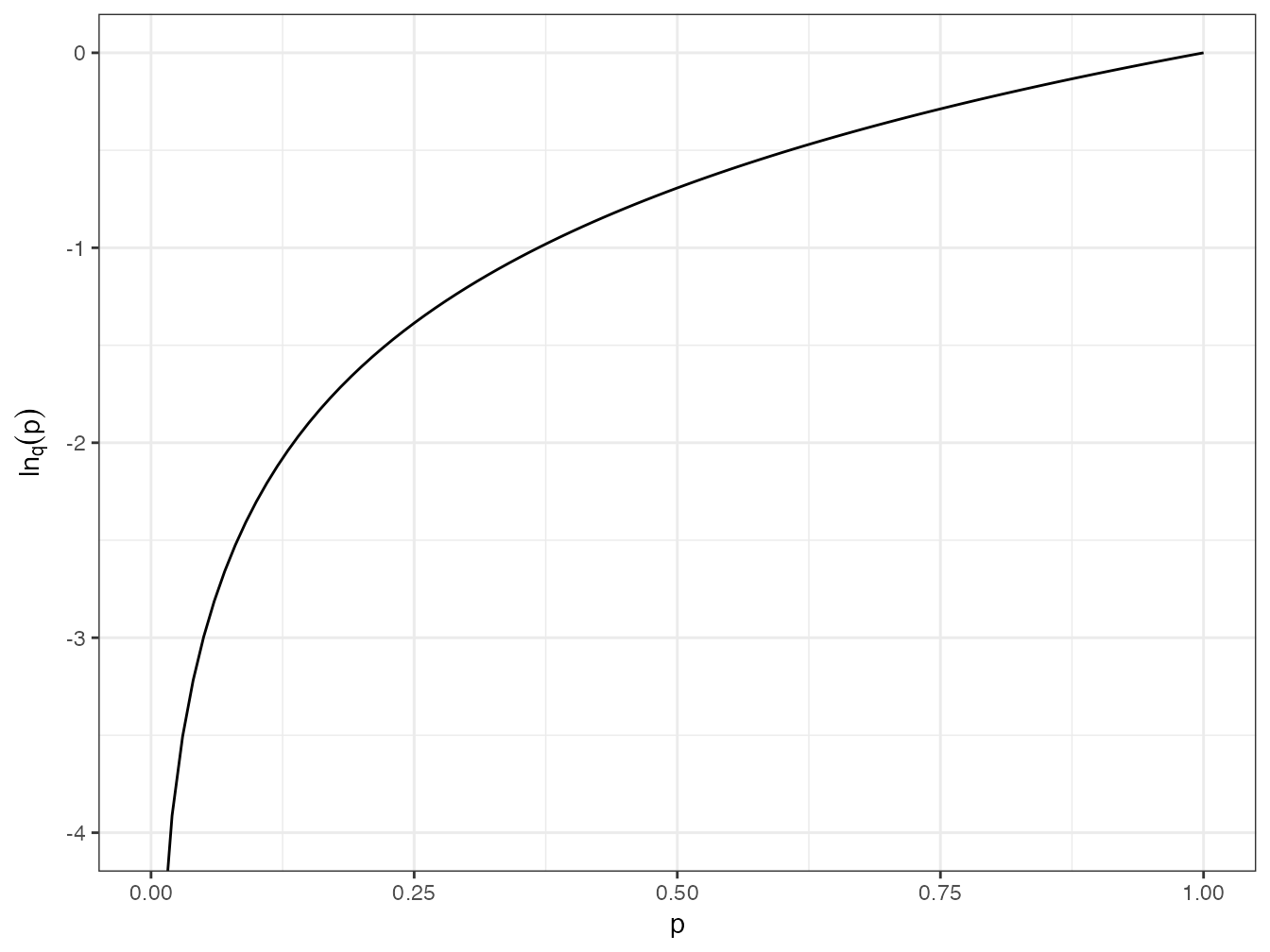
Figure 4.3: Valeur du logarithme d’ordre \(q\) de probabilités entre 0 et 1 pour différentes valeurs de \(q\) : \(q = 0\) (pointillés longs rouges), la courbe est une droite ; \(q = 1\) (trait plein) : logarithme naturel ; \(q = 2\) (pointillés courts bleus) : la courbe a la même forme que le logarithme naturel pour les valeurs positives de \(q\) ; \(q = -1\) (pointillés alternés verts) : la courbe est convexe pour les valeurs négatives de \(q\).
Le code R nécessaire pour réaliser la figure est :
ln0 <- function(p) lnq(p, 0)
ln2 <- function(p) lnq(p, 2)
lnm1 <- function(p) lnq(p, -1)
tibble(x = c(0, 1)) %>%
ggplot(aes(x)) +
stat_function(fun = log) +
stat_function(fun = ln0, lty = 2, col = "red") +
stat_function(fun = ln2, lty = 3, col = "blue") +
stat_function(fun = lnm1, lty = 4, col = "green") +
coord_cartesian(ylim = c(-4, 0)) +
labs(x = "p", y = expression(ln[q](p)))Sa fonction inverse est l’exponentielle d’ordre \(q\) :
\[\begin{equation} \tag{4.7} e^x_q = \left[ 1 + \left( 1 - q \right) x \right]^{\frac{1}{1 - q}}. \end{equation}\]
Enfin, le logarithme déformé est subadditif :
\[\begin{equation} \tag{4.8} \ln_q\left( xy \right) = \ln_q{x} + \ln_q{y} - \left( q - 1 \right) \left( \ln_q{x} \right) \left( \ln_q{y} \right). \end{equation}\]
Ses propriétés sont les suivantes :
\[\begin{equation} \tag{4.9} \ln_q\frac{1}{x} = -x^{q - 1} \ln_q{x}; \end{equation}\]
\[\begin{equation} \tag{4.10} \ln_q\left( xy \right) = \ln_q{x} + x^{1-q} \ln_q{y}; \end{equation}\]
\[\begin{equation} \tag{4.11} \ln_q\left( \frac{x}{y} \right) = \ln_q{x} - {\left( \frac{x}{y} \right)}^{1 - q} \ln_q{y}; \end{equation}\]
et
\[\begin{equation} \tag{4.12} e^{x + y}_q = e_q^x e_q^{\frac{y}{1 + \left( 1 - q \right) x}}. \end{equation}\]
Si \(q > 1\), \(\mathop{\lim}_{x \to +\infty}(\ln_q{x}) = {1} / {(q - 1)}\), donc \(e_q^x\) n’est pas définie pour \(x > 1 / (q - 1)\).
La dérivée du logarithme déformé est, quel que soit \(q\), \[\begin{equation} \tag{4.13} \ln'_q\left( x \right) = x^{-q}. \end{equation}\]
Les dérivées première et seconde de l’exponentielle déformée sont, quel que soit \(q\) : \[\begin{equation} \tag{4.14} \exp'_q\left( x \right) = \left( e_q^x \right)^{q}; \end{equation}\] \[\begin{equation} \tag{4.15} \exp''_q\left( x \right) = \left( e_q^x \right)^{2q - 1}. \end{equation}\]
Ces fonctions sont implémentées dans le package divent : ln_q(x, q) et exp_q(x, q).
L’entropie d’ordre \(q\) s’écrit
\[\begin{equation} \tag{4.16} ^{q}\!H = \frac{1}{q - 1} \left( 1 - \sum^S_{s=1}{p^q_s} \right) = -\sum_s{p^q_s} \ln_q{p_s} = \sum_s{p_s}{\ln_q\frac{1}{p_s}}. \end{equation}\]
Ces trois formes sont équivalentes mais les deux dernières s’interprètent comme une généralisation de l’entropie de Shannon (Marcon et al. 2014). La dernière est la plus intéressante parce qu’elle permet de définir l’entropie en général comme la moyenne du logarithme de l’inverse des probabilités, que nous appellerons la rareté des espèces.
Le calcul de \(^{q}\!H\) peut se faire avec la fonction ent_tsallis de la librairie divent :
ent_tsallis(bci_prob, q= 1.5)## # A tibble: 1 × 3
## estimator order entropy
## <chr> <dbl> <dbl>
## 1 naive 1.5 1.724.6 Entropie et diversité
On voit immédiatement que l’entropie de Tsallis est le logarithme d’ordre \(q\) du nombre de Hill correspondant, comme l’entropie de Rényi en est le logarithme naturel :
\[\begin{equation} \tag{4.17} ^{q}\!H = \ln_q{^{q}\!D}; \end{equation}\]
\[\begin{equation} \tag{4.18} ^{q}\!D = e_q^{^{q}\!H}. \end{equation}\]
L’entropie est utile pour les calculs : la correction des biais d’estimation notamment. Les nombres de Hill, ou nombres équivalents d’espèces ou nombres effectif d’espèces permettent une appréhension plus intuitive de la notion de biodiversité (Jost 2006). En raison de leurs propriétés, notamment de décomposition (voir le chapitre ??), Jost (2007) les appelle “vraie diversité”. Hoffmann and Hoffmann (2008) critiquent cette définition totalitaire et fournissent une revue historique plus lointaine sur les origines de ces mesures. Jost (2009) reconnaît qu’un autre terme aurait pu être choisi (“diversité neutre” ou “diversité mathématique” par exemple).
Dauby and Hardy (2012) écrivent “diversité au sens strict” ; Gregorius (2010) “diversité explicite”.
Quoi qu’il en soit, les nombres de Hill respectent le principe de réplication (voir Chao, Chiu, and Jost (2010), section 3 pour une discussion et un historique) : si \(I\) communautés de même taille, de même niveau de diversité \(D\), mais sans espèces en commun sont regroupées dans une méta-communauté, la diversité de la méta-communauté doit être \(I \times D\).
L’intérêt de ces approches est de fournir une définition paramétrique de la diversité, qui donne plus ou moins d’importance aux espèces rares :
- \(^{-\infty}\!D = {1} / {\min(p_S)}\) est l’inverse de la proportion de la communauté représentée par l’espèce la plus rare (toutes les autres espèces sont ignorées). Le biais d’estimation est incontrôlable : l’espèce la plus rare n’est pas dans l’échantillon tant que l’inventaire n’est pas exhaustif ;
- \(^{0}\!D\) est le nombre d’espèces (alors que \(^{0}\!H\) est le nombre d’espèces moins 1). C’est la mesure classique qui donne le plus d’importance aux espèces rares : toutes les espèces ont la même importance, quel que soit leur effectif en termes d’individus. Il est bien adapté à une approche patrimoniale, celle du collectionneur qui considère que l’existence d’une espèce supplémentaire a un intérêt en soi, par exemple parce qu’elle peut contenir une molécule valorisable. Comme les espèces rares sont difficiles à échantillonner, le biais d’estimation est très important, et sa résolution a généré une littérature en soi (section 3.1) ;
- \(^{1}\!D\) est l’exponentielle de l’indice de Shannon donne la même importance à tous les individus. Il est adapté à une approche d’écologue, intéressé par les interactions possibles : le nombre de combinaisons d’espèces en est une approche satisfaisante. Le biais d’estimation est sensible ;
- \(^{2}\!D\) est l’inverse de l’indice de concentration de Gini-Simpson donne moins d’importance aux espèces rares. Hill (1973) l’appelle “le nombre d’espèces très abondantes”. Il comptabilise les interactions possibles entre paires d’individus : les espèces rares interviennent dans peu de paires, et influent peu sur l’indice. En conséquence, le biais d’estimation est très petit ; de plus, un estimateur non biaisé existe ;
- \(^{\infty}\!D = {1} / {d}\) est l’inverse de l’indice de Berger-Parker (Berger and Parker 1970) qui est la proportion de la communauté représentée par l’espèce la plus abondante : \(d = \max(\mathbf{p})\). Toutes les autres espèces sont ignorées.
Le calcul de \(^{q}\!D\) peut se faire avec la fonction div_hill de la librairie divent :
div_hill(bci_prob, q = 1.5)## # A tibble: 1 × 3
## estimator order diversity
## <chr> <dbl> <dbl>
## 1 naive 1.5 49.6Les propriétés mathématiques de la diversité ne sont pas celles de l’entropie. L’entropie doit être une fonction concave des probabilités comme on l’a vu plus haut, mais pas la diversité (un exemple de confusion est fourni par Gadagkar (1989), qui reproche à \(^{2}\!D\) de ne pas être concave). L’entropie est une moyenne pondérée par les probabilités de la fonction d’information, c’est donc une fonction linéaire des probabilités, propriété importante pour définir l’entropie \(\alpha\) (section ??) comme la moyenne des entropies de plusieurs communautés, ou l’entropie phylogénétique (chapitre ??) comme la moyenne de l’entropie sur les périodes d’un arbre. La diversité n’est pas une fonction linéaire des probabilités : la diversité moyenne n’est en général pas la moyenne des diversités.
4.7 Synthèse
L’inverse de la probabilité d’une espèce, \(1/p_s\), définit sa rareté. L’entropie est la moyenne du logarithme de la rareté :
\[\begin{equation} \tag{4.19} ^{q}\!H = \frac{1}{q-1} \left( 1 - \sum^S_{s=1}{p^q_s} \right) = \sum_s{p_s}\ln_q\frac{1}{p_s}. \end{equation}\]
La diversité est son exponentielle :
\[\begin{equation} \tag{4.20} ^{q}\!D = e_q^{^{q}\!H}. \end{equation}\]
4.8 Estimation
Les estimateurs peuvent être classés dans quatre méthodes principales. La plus simple consiste simplement à insérer l’estimateur de \(p_s\) dans la définition de la diversité à évaluer pour obtenir ce que l’on appelle l’estimateur plug-in. Quand l’estimateur des probabilités est \(\hat{p}_s = n_s/n\), l’estimateur est dit naïf. L’estimateur naïf de l’entropie HCDT d’ordre \(q\) est :
\[\begin{equation} ^{q}{\hat{H}} = \sum_{s}{{\hat{p}}_s \ln_q{\frac{1}{\hat{p}}_s}} \tag{4.21} \end{equation}\]
L’estimateur naïf est inutile dans les communautés hyperdiverses car il sous-estime fortement la diversité en raison des espèces non observées et de la non-linéarité de la l’entropie en fonction des probabilités.
Des progrès ont été réalisés dans l’estimation de la distribution réelle de la probabilité des espèces en ajustant un modèle de leur distribution aux données. La distribution des espèces non observées peut être ajoutée si leur nombre est estimé et qu’une forme de distribution est choisie. La première méthode d’estimation de l’entropie consiste donc à estimer précisément la distribution réelle des probabilités puis à lui appliquer l’estimateur plug-in.
Chao et al. (2015) ont utilisé un modèle à deux paramètres basé sur l’estimation de la couverture généralisée de l’échantillon (non détaillé ici), estimé la richesse totale avec l’estimateur Chao1 et modélisé les espèces non observées comme une distribution géométrique pour dévoiler la distribution complète rang-abondance d’une communauté observée. Ils ont appliqué l’estimateur plug-in à cette distribution pour obtenir l’estimateur appelé “Chao-unveiled” dans divent. L’estimateur “iChao-unveiled” est une variation de l’estimateur “Chao-unveiled”, où la richesse est estimée par l’estimateur iChao1.
L’estimateur du jackknife (Burnham and Overton 1979) a montré de bonnes performances pour estimer la richesse lorsque l’effort d’échantillonnage est trop faible pour que l’estimateur Chao1 soit performant (Brose, Martinez, and Williams 2003).
L’estimation de la richesse à l’aide de l’estimateur du jackknife, dont l’ordre est choisi en fonction des données, définit l’estimateur “jackknife-unveiled”. L’utilisation de l’estimateur jackknife pour estimer la queue de la distribution d’abondance n’était pas l’intention de Chao et al. (2015) car elle n’est pas cohérente avec leur cadre théorique.
La seconde méthode repose sur l’estimateur Horvitz and Thompson (1952) de la somme pondérée d’une fonction de ses éléments \({x_1, x_2, ..., x_S}\), soit \(\sum_{s}{p_s f(x_s)}\) lorsque certains d’entre eux ne sont pas observés. Un estimateur sans biais de la somme est obtenu lorsque chaque terme est divisé par sa probabilité d’être observé \(1-(1-p_s)^n\). Chao and Shen (2003) ont proposé de le combiner avec l’estimateur de la couverture de l’échantillon : conditionnellement à l’ensemble des espèces observées, un estimateur sans biais (Ashbridge and Goudie 2000) de \(p_s\) est \(\tilde{p}_s = \hat{C} \hat{p}_s\). Chao et Shen ont estimé l’entropie de Shannon ; la méthode a ensuite été étendue à l’entropie HCDT (Marcon et al. 2014).
\[\begin{equation} ^{q}{\tilde{H}} =\sum_{s}{\frac{ \hat{C}{\hat{p}}_s \ln_q\dfrac{1}{\hat{C}{\hat{p}}_s} }{ 1 - {\left( 1 - \hat{C}{\hat{p}}_s \right)}^n} } \end{equation}\]
Un progrès supplémentaire peut être fait en remplaçant l’estimateur conditionnel des probabilités \(\tilde{p}_s = \hat{C} \hat{p}_s\) par celui de Chao et al. (2015). Étant donné que l’estimateur de probabilité amélioré dépend de la couverture généralisée de l’échantillon, l’estimateur amélioré de Chao-Shen est appelé “generalized coverage”.
La troisième méthode a été établie par Grassberger (1988) qui a donné un estimateur à biais réduit de la valeur d’un entier à la puissance \(q\). \({p_s^q}\) s’écrit \(n_s^q/n^q\) et \(n_s^q\) est estimé (Marcon et al. 2014) par :
\[\begin{equation} \tilde{n}^{q}_{s} = \frac{\mathrm{\Gamma}\left(n_s + 1 \right)}{\mathrm{\Gamma}\left( n_s - q + 1 \right)} + \frac{{\left( -1 \right)}^n \mathrm{\Gamma}\left( 1 + q \right)\sin{\pi q}}{\pi \left( n + 1 \right)} \end{equation}\]
L’estimateur de \({p_s^q}\) est simplement \(\tilde{p}^{q}_{s} = \tilde{n}^{q}_{s}/n^q\). On l’introduit dans la formule de l’entropie pour obtenir l’estimateur de Grassberger :
\[\begin{equation} ^{q}{\tilde{H}} = \frac{1 - \sum_{s}{\tilde{p}^{q}_{s}}}{q - 1} \tag{4.22} \end{equation}\]
La dernière méthode a fait l’objet d’une importante littérature. Une revue peut être trouvée dans Chao, Wang, and Jost (2013), Appendix A. Elle repose sur l’estimation de \(h_q = \sum_{s}{p_s^q}\). \(h_q\) peut être écrit comme la somme suivante :
\[\begin{equation} h_q = \sum_{r=0}^{\infty}{\binom{q - 1}{r} \left( -1 \right)^r \zeta_r} \tag{4.23} \end{equation}\]
\(\zeta_r\) est l’entropie de Simpson généralisée \(\sum_{s}{p_s(1 - p_s)^r}\) définie par Z. Zhang and Zhou (2010). Les \(n\) premiers éléments de la somme, notés \(\tilde{h}_{q}\), peuvent être estimés sans biais (Z. Zhang and Grabchak 2016) :
\[\begin{equation} \tilde{h}_{q} = \sum_{s=1}^{S}{\hat{p}_{s} \sum_{v=1}^{n - n_s}{\left[ \prod_{i=1}^{v}{\frac{i - q}{i} \prod_{j=1}^{v}{\left( 1 - \frac{n_s - 1}{n - j} \right)}} \right] }} \end{equation}\]
Z. Zhang (2013) montre que le biais dû à l’ignorance des termes restants est asymptotiquement normal et décroît exponentiellement vite. L’estimateur de Z. Zhang and Grabchak (2016) est celui basé sur \(\tilde{h}_{q}\) :
\[\begin{equation} ^{q}{\tilde{H}} = \frac{1 - \tilde{h}_{q}}{q - 1} \tag{4.24} \end{equation}\]
Des tentatives ont été faites pour estimer le biais restant (Z. Zhang and Grabchak 2013). La plus aboutie est celle de Chao and Jost (2015), qui complète Chao, Wang, and Jost (2013). Elle repose sur l’estimation du nombre total d’espèces par l’estimateur Chao1 et quelques approximations, notamment que les probabilités réelles des espèces non observées peuvent être supposées presque égales. Il en résulte que l’estimateur de la probabilité moyenne des espèces échantillonnées une fois est également égal à l’estimateur de la probabilité des espèces non observées. Sa valeur est notée \(A\). Elle vaut \(2 f_2 / [(n - 1) f_1 +2 f_2]\) si les singletons et les doubletons sont présents ou \(2 / [(n - 1)(f_1 -1) + 2]\) si les doubletons sont absents. L’estimateur Chao-Jost de l’entropie HCDT est :
\[\begin{equation} ^{q}{\tilde{H}} = \frac{1}{q - 1} \left[ 1 - \tilde{h}_q - \frac{f_1}{n}{\left( 1 - A \right)}^{1 - n} \left( A^{q - 1} - \sum_{r=0}^{n-1}{\binom{q - 1}{r} {\left( A - 1 \right)}^r} \right) \right] \end{equation}\]
En l’absence de singletons et de doubletons, \(A\) est fixé à 1 et l’estimateur est identique à celui de Zhang et Grabchak.
4.9 Profils de diversité
Leinster and Cobbold (2012), après Hill (1973), Patil and Taillie (1982), Tothmeresz (1995) et Kindt, Van Damme, and Simons (2006), recommandent de tracer des profils de diversité, c’est-à-dire la valeur de la diversité \(^{q}\!D\) en fonction de l’ordre \(q\) (figure 4.4) pour comparer plusieurs communautés. Une communauté peut être déclarée plus diverse qu’une autre si son profil de diversité est au-dessus de l’autre pour toutes les valeurs de \(q\). Si les courbes se croisent, il n’y a pas de relation d’ordre (Tothmeresz 1995).
Lande, DeVries, and Walla (2000) montrent que si la diversité de Simpson et la richesse de deux communautés n’ont pas le même ordre, alors les courbes d’accumulation du nombre d’espèces en fonction du nombre d’individus échantillonnés se croisent aussi.
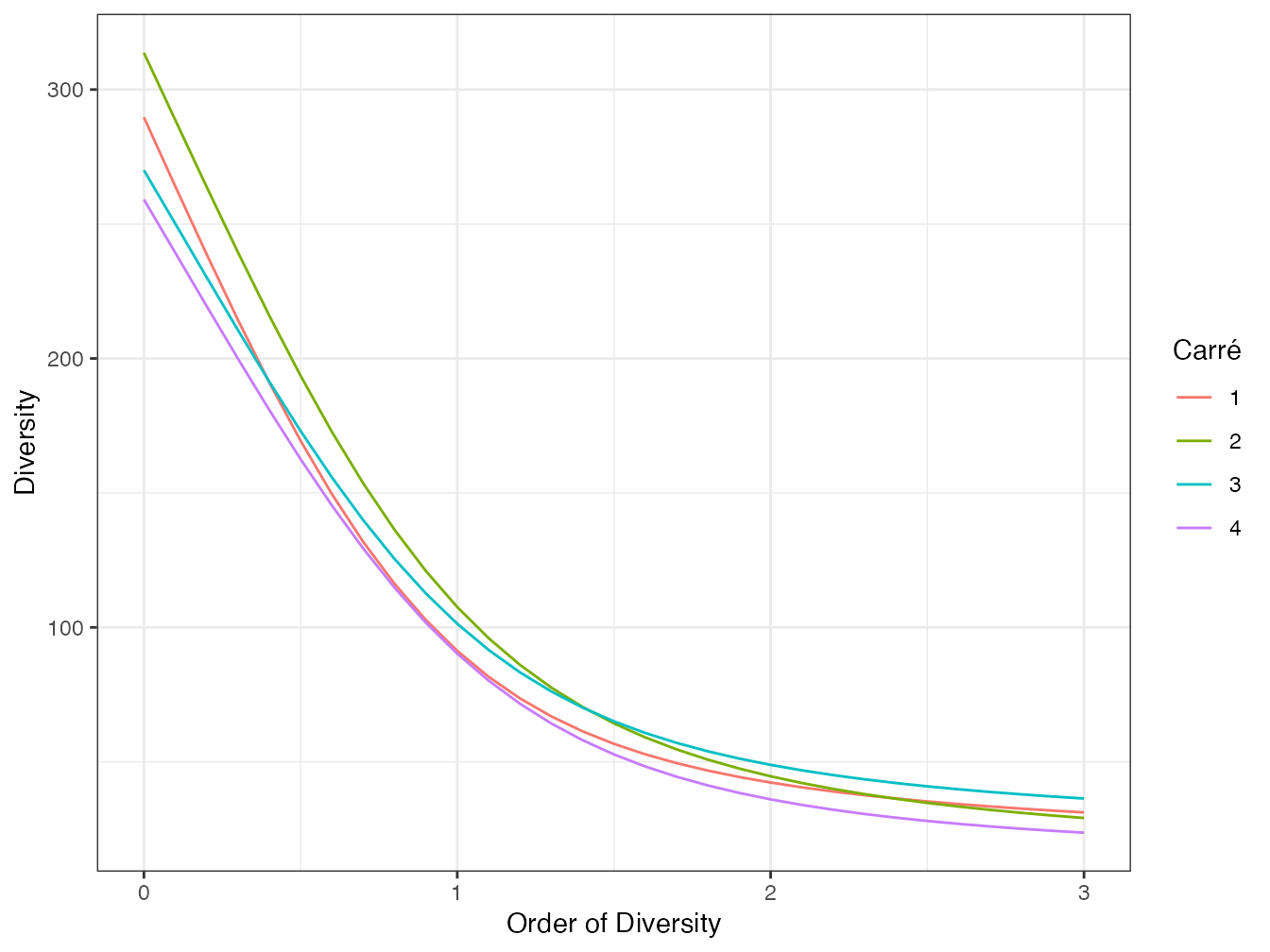
Figure 4.4: Profils de diversité des quatre carrés de la parcelle 6 de Paracou. La correction du biais d’estimation est celle de Chao et Jost. Le carré 2 est plus divers que le carré 4, mais pas que les carrés 1 et 3, qui sont plus divers dans les grands ordres de diversité. Le profil de diversité est tracé ici jusqu’à l’ordre \(q = 3\).
Code R pour réaliser la figure 4.4 :
paracou_6_abd %>%
# Suppression de "subplot_" dans les noms des carrés
mutate(site = str_replace(.$site, "subplot_", "")) %>%
# Profil
profile_hill(orders = seq(0, 3, .1), estimator = "ChaoJost") %>%
autoplot() +
labs(color = "Carré")Pallmann et al. (2012) ont développé un test statistique pour comparer la diversité de deux communautés pour plusieurs valeurs de \(q\) simultanément.
C. Liu et al. (2006) nomment séparables des communautés dont les profils ne se croisent pas. Ils montrent que des communautés peuvent être séparables en selon un profil de diversité de \(^{q}\!D\) sans l’être forcément selon un profil de diversité de Hurlbert (section 3.4), et inversement. Ils montrent que les communautés séparables selon un troisième type de profil, celui de la queue de distribution (Patil and Taillie 1982), le sont dans tous les cas. Le profil de la queue de distribution est construit en classant les espèces de la plus fréquente à la plus rare et en traçant la probabilité qu’un individu appartienne à une espèce plus rare que l’espèce en abscisse (figure 4.5).
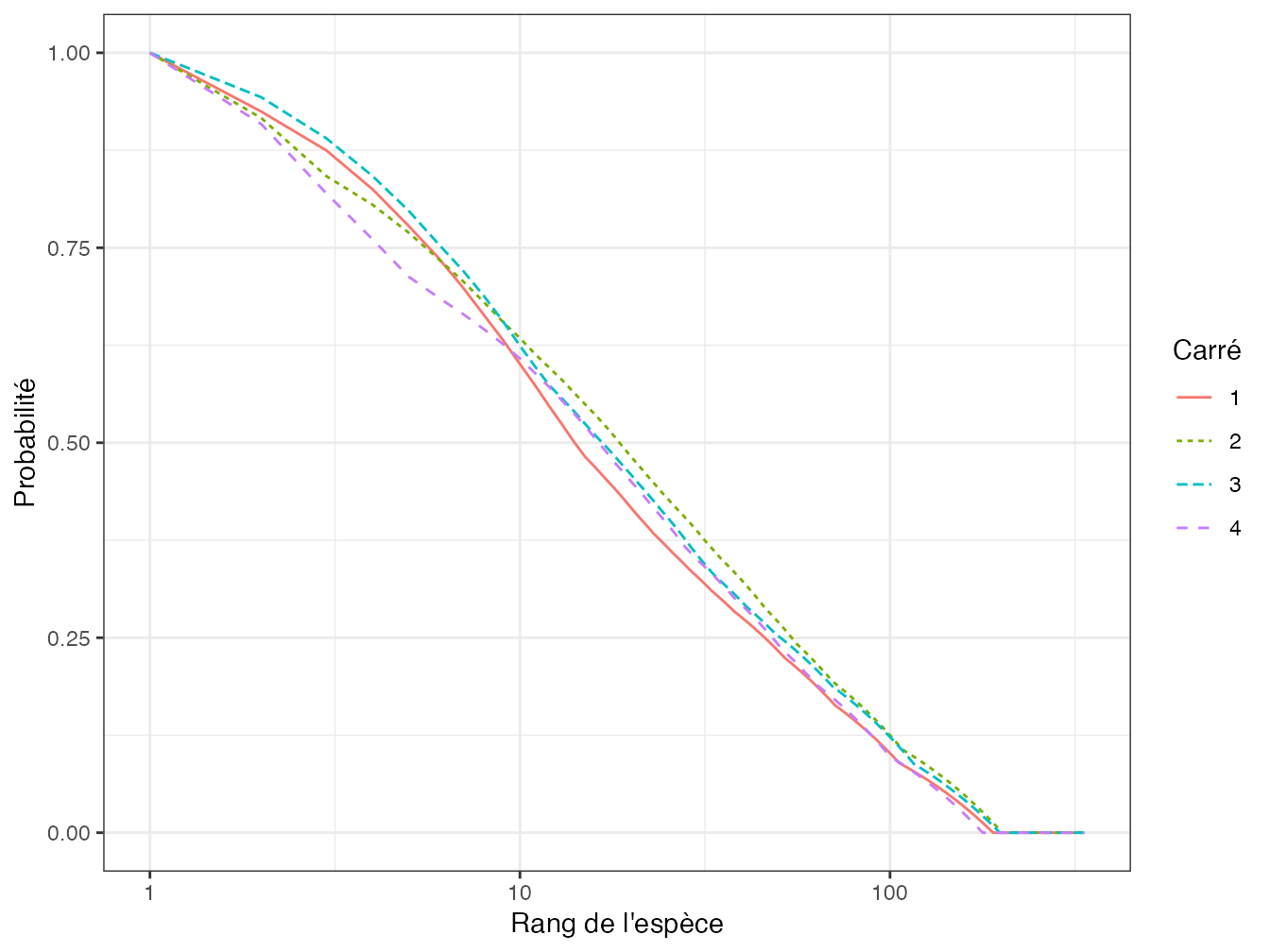
Figure 4.5: Profil de queue de distribution calculé pour les carrés de la parcelle 6 de Paracou. En abscisse : rang de l’espèce dans le classement de la plus fréquente à la plus rare ; en ordonnée : probabilité qu’un individu de la communauté appartienne à une espèce plus rare. Les profils de queue de distribution se croisent d’autant plus tôt qu’ils se croisent pour de grands ordres de diversité dans la figure 4.4.
Code R :
paracou_6_abd %>%
# Transformation en probabilités
as_probabilities() %>%
# Elimination des colonnes de description
as.matrix() %>%
# Tri des espèces par ordre croissant de probabilité
apply(MARGIN = 1, FUN = sort) %>%
# Cumul des probabilités. Attention: apply a transposé la matrice
apply(MARGIN = 2, FUN = cumsum) %>%
# Classement par valeurs décroissantes
apply(MARGIN = 2, FUN = rev) %>%
# Création d'un tibble pour le graphique
as_tibble() %>%
# Nom des colonnes: 1 à 4
rename_with(~ str_replace(., "V", "")) %>%
# Ajout d'une colonne pour l'ordre
mutate(rank = seq_len(nrow(.))) %>%
# Création du graphique
pivot_longer(cols = -rank) %>%
ggplot() +
geom_line(aes(x = rank, y = value, color = name, lty = name)) +
scale_x_log10() +
labs(
x = "Rang de l'espèce",
y = "Probabilité",
color = "Carré",
lty = "Carré"
) Les coordonnées des points du profil sont définies par \[\begin{equation} \tag{4.25} y(x) = \sum_{s=x+1}^{S}{p_{[s]}},\ x\in \{0, 1, \dots, S\}. \end{equation}\]
\(p_{[s]}\) est la probabilité de l’espèce \(s\) ; les espèces sont classées par probabilité décroissante.
Ce profil est exhaustif (toutes les espèces sont représentées) alors que les autres profils de diversité ne sont représentés que pour un intervalle restreint du paramètre et qu’un croisement de courbes peut se produire au-delà. En revanche, il ne prend pas en compte les espèces non observées.
Fattorini and Marcheselli (1999) proposent un test pour comparer deux profils de queue de distribution à partir d’échantillonnages multiples (nécessaires pour évaluer la variance de chacune des probabilités) mais qui néglige les espèces non observées.